Eine neue Methode macht neuronale Netze schlanker, effizienter und besser verständlich – mit Auswirkungen über die Fächergrenzen hinweg.

Jonas Fischer, Forschungsgruppenleiter „Explainable Machine Learning“ am Max-Planck-Institut für Informatik. Bild: Philipp Zapf-Schramm/MPI INF
Künstliche neuronale Netze treiben den wissenschaftlichen Fortschritt voran – etwa in der Medizin, wo sie helfen, genetische Muster zu entschlüsseln oder neue Therapieansätze zu entwickeln. Doch während diese Modelle immer komplexer werden, bleibt oft unklar, warum sie bestimmte Entscheidungen treffen – eine Herausforderung für Forschende, die damit neue Erkenntnisse gewinnen und bestehendes Wissen überprüfen wollen. Ein Team unter Beteiligung des Max-Planck-Instituts für Informatik hat nun eine Methode entwickelt, mit der neuronale Netze effizienter und interpretierbarer werden. Und zwar indem vorhandenes Fachwissen gezielt genutzt wird, um ihre Struktur zu optimieren.
Die Arbeit unter dem Titel „Pruning Neural Network Models for Gene Regulatory Dynamics Using Data and Domain Knowledge” verfasste der Saarbrücker Max-Planck-Forscher Jonas Fischer, Forschungsgruppenleiter zum Thema „Explainable Machine Learning“ in der Abteilung „Computer Vision and Machine Learning“, gemeinsam mit Inthekab Hossain und Professor John Quackenbush vom „Department of Biostatistics“ der Harvard T.H. Chan School of Public Health sowie Rebekka Burkholz, Tenure-Track Faculty am Saarbrücker Helmholtz Zentrum für Informationssicherheit Cispa und Leiterin der dortigen Forschungsgruppe „Relational Machine Learning“.
Ein großes Problem moderner neuronaler Netze ist die sogenannte „Überparametrisierung“: Modelle haben oft Millionen bis Milliarden von Parametern. Zwar sind solche Modelle in vielen Bereichen extrem leistungsfähig, doch ihre hohe Komplexität erschwert die Nachvollziehbarkeit – wie bekannte Large Language Models à la ChatGPT zeigen. In der Wissenschaft kann diese Intransparenz den Erkenntnisgewinn erheblich einschränken. Um dieses Problem zu lösen, können gezielt Netzwerkverbindungen entfernt werden, um Modelle schlanker zu machen (sog. „Pruning“).
Bisherige Pruning-Methoden zielten hauptsächlich auf die Hardware-Effizienz ab. Mit „Domain-Aware Sparsity Heuristic“ (DASH) haben die Forschenden eine Methode entwickelt, die Pruning gezielt zur Steigerung der Interpretierbarkeit nutzt. Dabei wird gesichertes Fachwissen einer Disziplin eingebunden, um das Modell bereits während des Trainings auf wissenschaftlich plausible Strukturen auszurichten: Nach einer Lernphase werden die entstandenen Gewichte mit Fachwissen abgeglichen, geprüft und unbrauchbare Verbindungen frühzeitig entfernt. Um auf Schwankungen in der Datenqualität oder Unsicherheiten im vorhandenen Expertenwissen reagieren zu können, haben die Forschenden eine Steuerungsvariable integriert: Damit kann flexibel gewichtet werden, ob beim Pruning stärker auf Expertenwissen, oder auf die während des Trainings gelernten Strukturen gesetzt wird. Dieser Prozess wird wiederholt, bis das Modell automatisch eine optimale Balance zwischen „Sparsity“ – also einer möglichst schlanken Netzwerkstruktur mit wenigen, aber relevanten Verbindungen – und Leistungsfähigkeit erreicht.
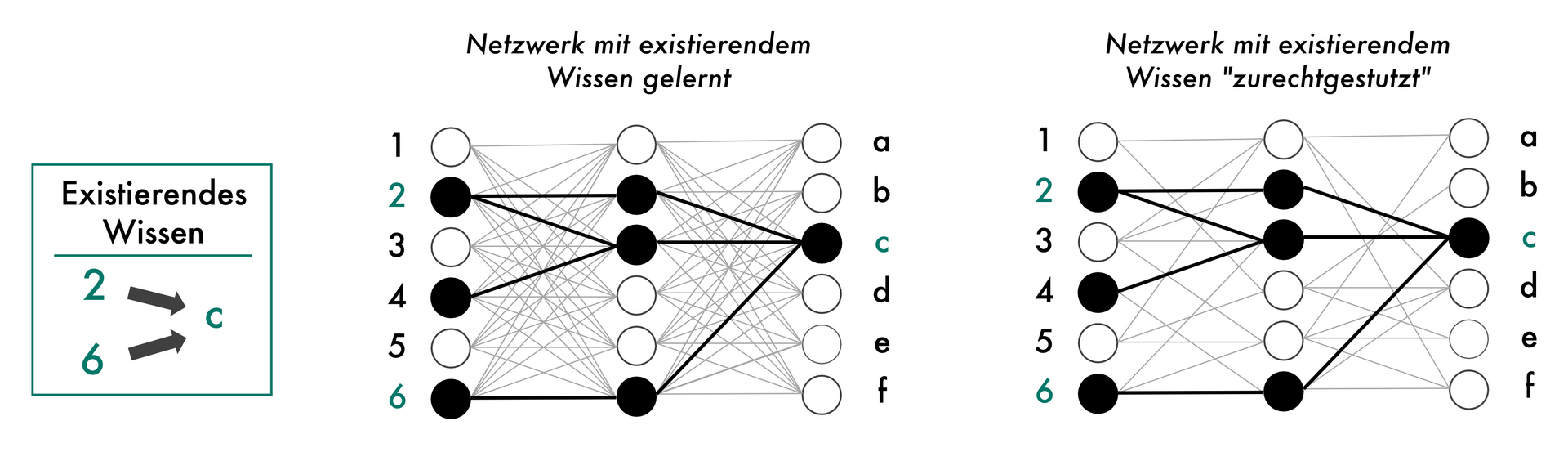
Um existierendes Wissen in einer Domäne für Neuronale Netze zu nutzen, können wir während des Lernprozesses das Neuronale Netzwerk dahingehend anpassen, dass das Wissen im Entscheidungsprozess widergespiegelt wird. Gerade in den Naturwissenschaften wie der Biologie ist solch ein Expertenwissen über Jahrzehnte aufgebaut und mehrfach im Labor bestätigt worden und sorgt so für robustere Entscheidungen. Unsere Methode erlaubt es uns nun auch anhand von existierendem Wissen das Netzwerk "zurechtstutzen", also unwichtige Verbindungen zu entfernen (rechts), wodurch Entscheidungen nachvollziehbarer und die Neuronalen Netzwerke auch leistungsstärker werden können. Urheber: Jonas Fischer
Die Forschenden haben ihre Methode zunächst auf den Bereich der Genregulation angewandt, da hier eine hohe Interpretierbarkeit der Modelle besonders wertvoll ist. „Die Erhebung von Genregulations-Daten ist extrem kostspielig. Doch neue Erkenntnisse auf diesem Gebiet können entscheidende Impulse für die Therapien schwerer Krankheiten wie Krebs und andere medizinische oder pharmazeutische Anwendungen liefern“, erklärt Jonas Fischer.
Ein wichtiger Aspekt von DASH ist deshalb die gezielte Wissensextraktion. Durch die Verbindung von maschinellem Lernen mit validiertem Fachwissen wird die Struktur der Modelle nachvollziehbarer, was es Fachleuten erleichtert, Zusammenhänge in den Netzen zu untersuchen. „Unsere Methode könnte helfen, biologisch relevante Muster sichtbar zu machen, die in überparametrisierten oder rein auf Effizienz optimierten Modellen untergehen“, so Fischer. Die in den gelernten Gewichten enthaltenen regulatorischen Muster ließen sich mit experimentellen Methoden weiter analysieren und auf ihre wissenschaftliche Relevanz hin überprüfen.
In ihrer Studie testeten die Forschenden DASH sowohl auf synthetischen Daten als auch auf realen Datensätzen zum Zellzyklus in Hefe und Brustkrebs beim Menschen, wobei sie experimentelle Goldstandard-Daten als Referenz verwendeten. Sie zeigten, dass ihre Methode vorangegangene Pruning-Ansätze deutlich übertrifft – sowohl in der wissenschaftlichen Aussagekraft der resultierenden Modelle, als auch in Hinblick auf deren Dateneffizienz und Leistungsfähigkeit. So konnten sie anhand des Modells einen signifikanten Zusammenhang zwischen dem Heme-Signalprozess und Veränderungen im Brustkrebs herstellen. Dieser Signalprozess wurde bereits früher mit einer Anti-Tumor Eigenschaft in Verbindung gebracht. Besonders interessant ist, dass zwei neue Regulatoren für diesen Signalprozess im Modell sichtbar wurden, die als Wirkstoffziel für Medikamente nutzbar sein könnten. Für einen dieser Regulatoren existiert bereits ein Mittel, das nun möglicherweise bei der Brustkrebsbehandlung zum Einsatz kommen könnte („drug repurposing“).
Das Team geht davon aus, dass ihre Methode auch auf andere Disziplinen übertragbar ist und eine Blaupause für eine neue Art der Optimierung neuronaler Netze liefern könnte – insbesondere in den Naturwissenschaften, wo bereits hochwertiges, etabliertes Fachwissen existiert. Die Forschenden haben gezeigt, dass ihre Methode selbst bei unvollständigem oder leicht fehlerhaftem Fachwissen noch zuverlässige Ergebnisse liefert. Eine Einschränkung: „Stark fehlerhaftes Expertenwissen beeinträchtigt die Qualität der von DASH erzeugten Modelle“, erklärt Jonas Fischer.
Die Arbeit veröffentliche das Team bei der letzten Auflage der „Conference on Neural Information Processing Systems“ (NeurIPS), die Ende Dezember 2024 in Vancouver, Kanada, stattfand. NeurIPS ist eine der weltweit führenden Fachkonferenzen im Bereich der Künstlichen Intelligenz, mit besonderem Fokus auf neuronale Netze. Zudem wurde Vorgängerarbeit des Teams zu der nun veröffentlichten Studie vom amerikanischen National Cancer Institute (NCI), der führenden Bundesbehörde und Forschungsorganisation für Krebsforschung in den USA, als eine wichtige Erkenntnis im Bereich der Krebsforschung Im Jahr 2024 hervorgehoben.
Originalpublikation:
I. Hossain, J. Fischer, R. Burkholz, J. Quackenbush (2024): „Pruning neural network models for gene regulatory dynamics using data and domain knowledge“. 38th Conference on Neural Information Processing Systems (NeurIPS).
https://proceedings.neurips.cc/paper_files/paper/2024/hash/d52d2281babd36913643392a09a56832-Abstract-Conference.html
Wissenschaftlicher Ansprechpartner:
Dr. Jonas Fischer
Max-Planck-Institut für Informatik
E-Mail: jonas.fischer[at]mpi-inf.mpg.de
Redaktion:
Philipp Zapf-Schramm
Max-Planck-Institut für Informatik
Tel: +49 681 9325 5409
E-Mail: pzs@mpi-inf.mpg.de
